Par David Larousserie Publié le 18 octobre 2021
ENQUÊTE La société DeepMind, filiale de Google, a mis au point un programme qui établit avec une justesse inégalée la structure tridimensionnelle des protéines. Une révolution conceptuelle qui va accélérer la recherche biomédicale.
Cette fois, c’est du sérieux. L’intelligence artificielle rivalise désormais avec les plus grands cerveaux scientifiques. Plus fort que les victoires dans les jeux de go, d’échecs, de poker, ou dans le jeu vidéo StarCraft. Moins anecdotique que les systèmes de reconnaissance de museaux de chats ou de chiens. Plus précis que les traductions automatiques fluides mais qui dérapent parfois. Moins risqué que les générateurs de fausses images, de fausses vidéos, ou d’œuvres d’art « à la manière de… ». La nouvelle vague de l’intelligence artificielle, celle des réseaux de neurones et de l’apprentissage statistique, vient de résoudre pour la première fois un important problème scientifique, digne d’un Nobel.
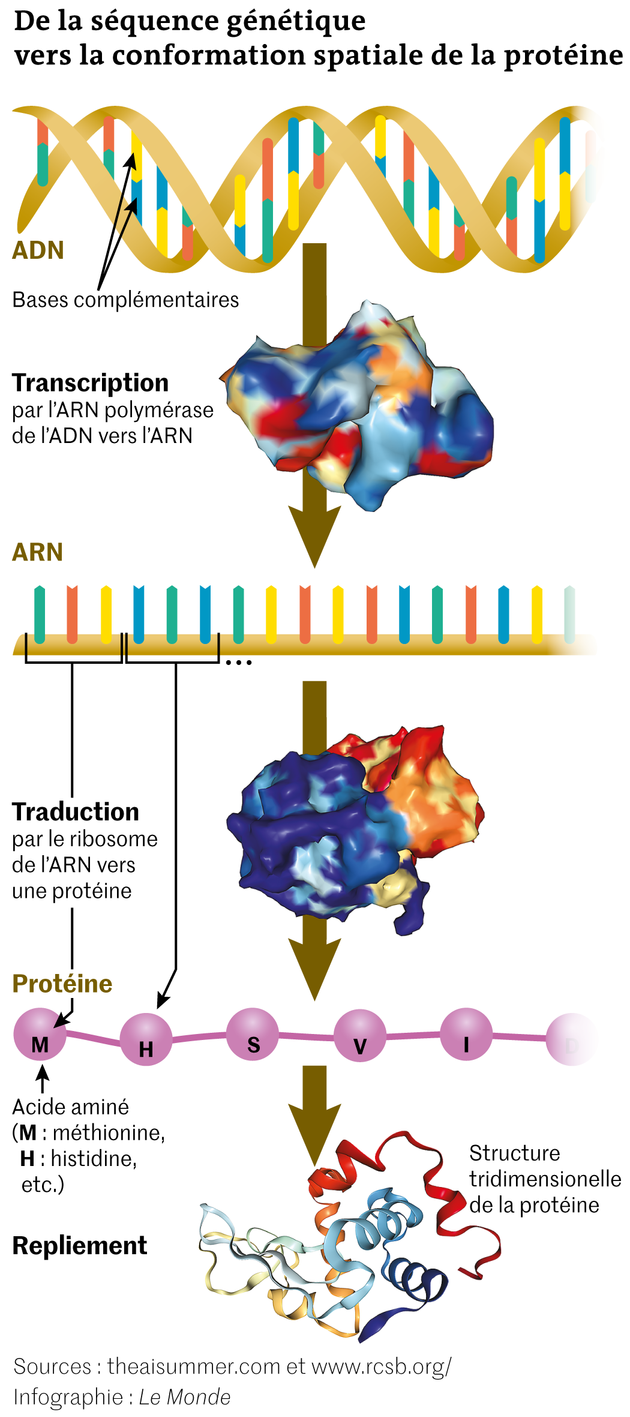
C’est d’ailleurs le prix Nobel 1972 de chimie, Christian Anfinsen, qui posa les bases du problème en question, central en biologie moléculaire. Comment se fabriquent les protéines, ces incroyables machines du vivant comme les anticorps, les enzymes, certaines hormones, l’hémoglobine, ou par exemple le SARS-CoV-2 (qui en compte vingt-neuf)… ? Comment passe-t-on du plan de montage, succession des lettres du génome, à une pièce complète et complexe de milliers d’atomes, capable de ressembler à une hélice, un marteau, un crochet, un tube, un marcheur sur deux jambes, des ciseaux…, pour ne citer que quelques formes associées à certaines des 20 000 de ces molécules que contient l’organisme humain ? En termes plus biologiques, comment ces protéines, sortes de long collier de petites molécules, des acides aminés, finissent-elles par se replier, se mettre en boule et adopter ces formes tridimensionnelles bien précises ? A quelles règles d’origami joue finalement la Nature ?
La bonne clé pour la bonne serrure
Ces questions en rafale n’ont rien d’anodin, car de la forme prise dans l’espace par ces spaghettis moléculaires, faits de centaines d’acides aminés, dépend la fonction de ces macromolécules.
Pour le comprendre, l’image de la clé correspondant à la bonne serrure s’impose. Une molécule odorante n’active un récepteur que si celui-ci est « accueillant » : un cube ne se logera pas dans une pyramide. Un anticorps ne s’accroche à un virus que s’il a le bon « crochet » pour s’y arrimer. Un neurotransmetteur ne réveille un neurone que si celui-ci s’ouvre, puis se ferme… A l’inverse, si la machinerie déraille et fabrique des molécules tordues, de même formule chimique que l’originale mais de formes légèrement différentes, alors cela peut dégénérer en maladie, comme dans celle de Creutzfeldt-Jakob.
Si bien que, pour avancer, les scientifiques sont obligés de connaître la forme adoptée par l’objet de leurs recherches, qu’ils soient biologistes pour saisir le rôle d’une protéine, ingénieurs en pharmacie pour trouver le bon médicament ciblant une molécule, ou chimistes pour concevoir un catalyseur accélérant une réaction… Sinon, ils restent dans le noir.
En cinquante ans, des progrès ont été faits – ne serait-ce qu’expérimentalement avec des techniques d’imagerie par rayonnement X ou par cryomicroscopie électronique – pour sortir de l’obscurité. Mais la théorie et la modélisation de ce repliement mystérieux patinaient jusqu’à l’avènement du programme informatique baptisé AlphaFold, développé par une filiale anglaise de Google, DeepMind.
« Je n’y croyais pas ! », se souvient John Moult, professeur à l’université du Maryland, fondateur en 1994 d’une compétition scientifique originale destinée à comparer les outils de repliement de protéines. Cette « évaluation critique de la prédiction de structures » (Critical Assessment of Protein Structure Prediction (CASP), en anglais) demande à un jury d’évaluer la qualité des prédictions de programmes testés sur une centaine de protéines dont la structure a été déterminée expérimentalement, mais n’a pas été encore publiée.
« Fin 2020, pour la quatorzième édition de CASP, on évaluait un à un les résultats du groupe 427. C’était parfait. Puis le second. Encore correct. Puis le troisième. Pareil. On allait d’étonnement en étonnement. C’était du solide », complète le spécialiste. Pour plus de 90 protéines, AlphaFold 2, le nom du programme du groupe 427, propose un résultat quasi parfait, donnant la position des éléments de la molécule à une précision atomique, aussi bonne que l’imagerie. « C’est fantastique ! Cinquante ans qu’on attendait cela. Le problème classique du repliement est résolu », apprécie John Moult. Le programme fait environ 50 % de mieux que son prédécesseur, AlphaFold 1, qui deux ans plus tôt avait déjà remporté la compétition. « C’est une grande percée », salue un autre pionnier, David Baker (université de Washington), arrivé deuxième avec un score deux fois plus faible que son concurrent.
Un usage démocratisé
C’était le 30 novembre 2020. Sept mois et demi plus tard, les 15 et 22 juillet 2021, DeepMind ajoute encore au plaisir de la communauté scientifique. L’entreprise publie dans Naturedeux articles de recherche détaillés sur sa méthode et met à disposition le code source de son programme. Puis, en partenariat avec le Laboratoire européen de biologie moléculaire (EMBL), elle met en ligne une base de données de plus de 350 000 structures de protéines, presque deux fois plus que la vénérable Protein Data Bank, référence du domaine, créée il y a cinquante ans. Plus de 98 % des protéines humaines y sont présentes, complétées de celles de la mouche, du poisson-zèbre, de levures…, soit vingt espèces au total.
DeepMind promet prochainement cent millions de configurations tridimensionnelles, soit la moitié environ des séquences d’acides aminés actuellement recensées dans la base UniProt. De quoi espérer combler le fossé entre le grand nombre de séquences connues et celui, bien plus faible, des structures résolues.
C’est une deuxième folie.
Sur Twitter, durant l’été, des équipes postent leurs premières structures. L’une évoque son succès à obtenir les modèles de douze protéines sur lesquelles elle travaillait depuis six ans. Le groupe de Sergey Ovchinnikov, à Harvard, met en ligne en moins d’un mois un notebook, un outil permettant de tester AlphaFold 2 sans installation sur son ordinateur, pour en démocratiser l’usage. « Il est même plus rapide que le notebook de DeepMind », constate Carlos Outeiral, doctorant de l’université d’Oxford qui, dès le 19 juillet, décortique sur son blog les secrets d’AlphaFold 2. Plus anecdotique, un site permet d’entrer ses nom et prénom dont les lettres forment une séquence d’acides aminés pour voir à quoi ressemble la protéine associée. « La période est excitante. Plein d’initiatives apparaissent », constate Sophie Sacquin-Mora, du laboratoire de biochimie théorique à l’Institut de biologie physico-chimique et autrice du blog de portraits de protéines « Top of the prots ». « J’ai rapidement essayé le programme sur des protéines dont il avait fallu des mois pour obtenir la structure. Et ça colle. C’est bluffant », témoigne Benjamin Bardiaux, à l’Institut Pasteur. Plus de 200 prépublications sur le site bioRxiv mentionnent déjà AlphaFold.
DeepMind a annoncé des partenariats avec l’organisation internationale de recherche Drugs for Neglected Diseases Initiative pour le développement de thérapies sur des maladies négligées, et avec le britannique Centre for Enzyme Innovation pour la recherche sur des procédés de recyclage du plastique.
Trois leviers : les données, le calcul et l’algorithmique
Le même jour que la publication de Nature, la revue concurrente, Science, publiait un algorithme concurrent, RoseTTAFold, du laboratoire de David Baker, développé en quelques mois sur les idées d’Alphafold, disponibles partiellement fin 2020. Cela a permis aux chercheurs de mettre au point et de fabriquer une protéine n’existant pas dans la nature et qui agrège en sphère plusieurs anticorps afin de doper leur action. Ils pensent ainsi rendre plus efficaces des vaccins anti-Covid-19. « Pour cette activité de design de protéines nouvelles, les choses s’accéléreront avec AlphaFold », prédit Stéphane Téletchéa, enseignant-chercheur à l’université de Nantes.
« Il est trop tôt pour anticiper les changements qu’apportera AlphaFold mais, grâce à ce programme, des choses impossibles deviennent possibles. Il fallait plusieurs mois pour avoir une structure alors que, là, quelques minutes ou heures suffisent »,note Carlos Outeiral.
Pour parvenir à ses fins, AlphaFold a eu recours aux trois leviers classiques de l’intelligence artificielle : les données, les ressources en calcul, l’algorithmique. Comme il s’agit d’un système d’apprentissage, DeepMind a puisé dans plusieurs bases de données existantes, l’une recensant des structures (Protein Data Bank), l’autre collectant des séquences (UniProt), pour apprendre les « règles » de construction d’un assemblage.
La ressource en calcul, elle, avait limité au début des années 2000 l’essor de ces techniques nées à la fin des années 1980. Cent vingt-huit processeurs spécifiques, issus des technologies de Google (les Tensor Processing Unit, équivalents des processeurs ou des cartes graphiques des ordinateurs), ont tourné plusieurs semaines, sans compter les nombreux prototypes nécessaires avant de tomber sur le meilleur. Enfin, les ingénieurs ont réuni plusieurs algorithmes d’intelligence artificielle issus du monde de l’imagerie ou de la compréhension du langage, d’une manière plus efficace que leurs concurrents. « Quatre ans d’efforts et un noyau de trente personnes ont été nécessaires »,indique un porte-parole de DeepMind.
A l’enthousiasme unanime se mêlent tout aussi vite les premiers bémols. « C’est une avancée majeure, mais il faut nuancer un peu l’engouement car c’est surtout un aboutissement pour lequel les bases étaient jetées depuis de nombreuses années », rappelle Isabelle Callebaut, chercheuse CNRS à l’Institut de minéralogie, de physique des matériaux et de cosmochimie de Sorbonne-Université. « Nous savions que cela arriverait, mais pas quand. Les idées étaient dans l’air », complète Stéphane Téletchéa.
Un trop bon élève
« Il faut rester prudent sur les résultats car il y a parfois des erreurs. J’ai pu constater que, pour une partie d’une protéine, le programme prédisait une région sans forme précise, alors qu’elle existe », pointe Carlos Outeiral. Il faudra donc sûrement toujours passer par une étape de vérifications expérimentales, plus longue, pour confirmer les conformations spatiales prédites.
« Il n’est pas tout à fait exact de dire que AlphaFold résout le problème du repliement des protéines, car le programme n’indique pas, par exemple, les étapes du repliement, ou pourquoi dans certains cas une protéine ne se replie pas »,modère Benjamin Bardiaux. Ce repliement se fait naturellement sous l’effet des différentes forces chimiques entre parties électriquement chargées, ou entre certains atomes… Et ces détails, utiles pour saisir la dynamique d’un repliement, sont encore l’objet de nombreux travaux.
Assez vite aussi, des faiblesses plus précises ont été décrites. AlphaFold est un bon élève. Il ne reculera pas devant l’obstacle et trouvera toujours une conformation (sur laquelle il fournit en plus une estimation de confiance), même si elle n’existe pas ! Or beaucoup de protéines, ou du moins des parties, n’ont en fait pas de structure et restent désordonnées. Elles ont même été baptisées « intrinsèquement désordonnées » et pourraient représenter jusqu’à 30 % du protéome des eucaryotes (les cellules à noyau, comme chez l’homme). Elles posent donc évidemment des problèmes insolubles à AlphaFold.
Autre écueil, l’outil fournit la forme repliée d’une protéine seule. Or cette forme est rarement observée dans la nature. Une protéine est en effet souvent associée soit à une autre (un anticorps à la surface d’un virus, un virus sur un récepteur de cellules), soit elle interagit avec de plus petites molécules, souvent appelées ligands, comme des ions, des métaux ou des peptides (faits de quelques acides aminés seulement). Soit elle est elle-même l’assemblage plus ou moins stable de plusieurs protéines, comme dans les usines énergétiques que sont les ATPase, ou les fabriques que sont les ribosomes, ou encore les virus… Tous ces mariages, appelés complexes, restent mal décrits par AlphaFold, entraîné sur les cas simples de molécules isolées.
Sans compter que ces complexes sont eux-mêmes en évolution, adoptant une forme ou une autre selon leur fonction. « La structure n’explique pas tout de la fonction d’une protéine, mais sans elle on ne comprendrait pas »,rappelle David Stroebel, chercheur CNRS à l’Institut de biologie de l’Ecole normale supérieure à Paris, qui cite l’exemple d’un récepteur neuronal qu’il étudie, capable d’adopter plusieurs conformations selon qu’il est lié ou non à un neurotransmetteur, qu’il est actif ou non. « Parmi ces quatre-là, quelle configuration donne AlphaFold ? »,s’interroge le chercheur.
Un nouveau modèle de recherche
Les ingénieurs de DeepMind se sont cependant déjà mis au travail sur ces épineuses questions. Le 4 octobre, ils ont même déposé une prépublication qui commence à régler le cas des interactions entre protéines. « La quinzième édition de CASP prévoit un volet “complexe” pour tester les nouvelles idées des scientifiques », annonce John Moult, mais DeepMind n’a pas encore confirmé sa participation à ce concours prévu pour 2022.
Derrière ces incontestables succès, certains soulignent une autre révolution, moins technique celle-là. Il s’agit de celle du modèle de recherche et du fait qu’une entreprise privée soit à l’origine de la percée. Certes, DeepMind n’est pas la première à illuminer la science puisque, avant elle, IBM a inventé le microscope à effet tunnel et découvert les supraconducteurs à haute température, et que les capteurs CCD des caméras sortent des Bell Labs, qui ont aussi observé le fond diffus cosmologique. Mais « nous voyons les prémisses d’un changement de paradigme. (…) Si la recherche publique mondiale était demain éclipsée par des laboratoires privés, les résultats des recherches les plus avancées passeraient de leur statut actuel de bien commun accessible à tous à celui de propriété intellectuelle de grands groupes, américains ou asiatiques », alerte le biologiste Patrick Lemaire, dans une tribune parue dans Le Monde le 11 octobre. Il y note qu’« en 2020, les entreprises Amazon, Google et Huawei ont, à elles seules, investi plus de 70 milliards d’euros en recherche et développement, soit environ une fois et demie l’investissement total, public et privé, de la France ».
« Ce n’est pas qu’une question de moyens humains ou de calculs. Le travail de DeepMind doit nous faire aussi réfléchir sur l’organisation de notre recherche. Ils ont réussi à faire collaborer ensemble pendant longtemps des gens avec des compétences différentes. Cela devrait inspirer le monde académique », témoigne Carlos Outeiral. « On devrait sans doute plus collaborer entre nous pour arriver à de telles réussites », complète John Moult. Comme des protéines complexes, le monde académique va-t-il changer de conformation ?


Aucun commentaire:
Enregistrer un commentaire